随着我国医院信息化水平的不断提高, 积累了大量的临床数据, 如何有效利用这些数据成为目前数据科学领域关注的重点之一。根据我国在2016年发布的57项电子病历相关的卫生行业标准[1], 涉及电子病历数据的共53项, 这53项电子病历数据标准中, 规定“ 人读部分” [示例见《电子病历共享文档规范第34部分:入院记录》(WS/T 500.34-2016)主诉章节的文本元素(text, 人读部分, P8)、现病史章节的文本元素(text, 人读部分, P9)等]的纯文本数据项共307处, 这些非结构化数据内蕴含了非常丰富的诊疗过程信息, 但其中信息的准确、全面提取则较为困难。
在整个电子病历数据集中, 就住院患者的电子病历数据而言, 尤以入院记录、病程记录、手术记录、出院小结等数据中包含的信息最为丰富。经考察目前我国已实施电子病历的医院, 上述4项数据所包含的具体信息如表1所示, 在实际系统实现策略不尽相同, 可能与表1略有出入, 但基本内容大体类似。
| 表1 电子病历文档中非结构化文本数据的相关章节取样列表 Table 1 Unstructured episodes in electronic medical records of China electronic medical records standards |
从数据的组织形式考察, 电子病历中涉及诊疗细节的很多信息, 例如主诉、现病史、既往史、鉴别诊断、影像诊断、手术记录等主体内主要采用中文自然语言文字描述, 是临床医生实际诊疗细节的具体体现, 包含了大量丰富信息的同时, 由于其内容与医生在整个诊疗过程中的思考路径密切关联, 很难做到基于统一的、严格的表格形式来组织, 因此在数据使用时就需要根据具体的数据实况进行定制化信息提取。由于这些非结构化的文本信息蕴含的价值丰富, 但处理、利用困难, 因此目前逐渐成为国际医学数据分析利用的热点[2]。
本研究以糖尿病病史的抽取与结构化为例, 讨论电子病历非结构化数据处理的自然语言处理(natural language processing, NLP)、文本挖掘等方法与算法, 以期为中文电子病历中的信息抽取提供方法参考。
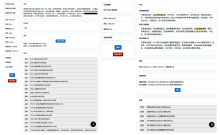
图1为包含病史信息的入院记录以及我们进行信息提取的需求示意图。就我们所了解的情况, 目前中文电子病历中, 疾病史的描述一般采用“ [疾病名][时间描述]” , 常见的描述有“ 糖尿病史20年” “ 糖尿病12年” “ 3年前患糖尿病” 等。这种变形在实际病历的书写中, 可能出现非常多的变化, 以本研究所选取的语料库标注的结果为例, 糖尿病史的叙述就出现了至少10种描述, 表2是出现频繁的前3个模式示例。
| 表2 糖尿病史的描述模式示例 Table 2 Text samples of diabetes history in electronic medical records |
对于给定的疾病, 在选定的电子病历数据集合基础上, 我们需要解决以下几个问题。
1.2.1 找到所有的糖尿病史描述句型(或模版) 如表2所示, 将电子病历数据中所有糖尿病史的叙述都找到, 按照类别进行归纳并总结为模版, 以便最终抽取所有的病史。这其中要解决的关键问题有两个:找到所有的时间表述以及所有的糖尿病确诊表述, 疾病确诊及其关联的病史时间表述。
1.2.2 抽取所有糖尿病患者的病史描述信息并组织为关系表 对于不同的模版, 编写统一的抽取规则, 以便将抽取的结果存储到结果表中去。重点需要解决针对模版进行规则重写的准确性、一致性和有效性。
1.2.3 验证抽取结果的准确性、全面性 对于抽取的结果, 需要确定其与数据中所记录的情况相符合的程度。由于实践中一般不可能实现全面的人工专家核对, 因此采取适当的抽检方法, 辅以自动化的检验工具来对抽取结果进行正确性、准确率、召回率的验证与计算是需要重点关注的问题。
采用如图2所示的处理流程实现糖尿病史的抽取与组织以及结果验证。
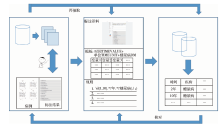 | 图2 非结构化电子病历文本数据中糖尿病史的抽取与组织Figure 2 Information extraction and curation of diabetes history in unstructured episodes |
图2共由5部分组成, 第一步是建立病史语料库, 通过3组共6人(文本标注每组2人, 共两组; 标注结果核对1组, 共2人)并行工作, 对语料库按照规范定制化标注, 目的是全面排查并分析得出糖尿病史描述的模版; 第二步是将前一步结果重写为特定语言的抽取规则库; 第三步是在电子病历非结构化数据中, 按照规则库对病史进行内容抽取, 并将结果存储到结果表格中; 第四步是对电子病历中的否定描述进行抽取并从前一步结果中排除可能的假阳性结果; 最后一步是采用人工抽检与自动化检验相结合的方式进行结果验证。
为了完整地从非结构化电子病历中抽取病史信息, 首先需要确定特定疾病病史(例如糖尿病病史)的代表性叙述都有哪些, 从而发现可能的规律并构造等价的抽取规则。我们将电子病历数据中病史部分(包括现病史、既往史、个人史等信息)作为语料库抽样的候选集, 针对病史抽取的定制化需求, 将所有含时间单位描述(年、月、日、天)的数据全部抽取为基础语料库, 接下来对基础语料库按医院、科室进行分组并按5%的量进行抽样, 这些抽样的数据作为标注候选集进入标注平台, 由经过培训的标注人员对这些候选集进行标注。
为了提高标注工作的效率, 我们实现了标注模版的在线学习及实时应用功能。例如标注人员标注以下结果:例1:“ 20年前发现患糖尿病” — — 20, TIMEVALUE; 年, TIMEUNIT; 前, TIMEPOS; 糖尿病, DM。例2:“ 糖尿病病史13年” — — 糖尿病, DM; 13, TIMEVALUE; 年, TIMEUNIT。若标注人员已经发现这种叙述在语料库中很常见, 可以通过平台提供的“ 模版化并应用” 的功能, 该功能首先将上述标注抽象为模版(注意[* ]表示可能的叙述, 可以为空) TIMEVALUE [* ] TIMEUNIT [* ] DM, 并以此模版对应的抽取规则为依据, 对尚未标注的语料库中的文本进行查询并自动标注, 其后标注人员需要对这些自动标注的结果进行确认后即可作为正式标注结果, 图3是标注平台的手工标注以及模版化应用的示意图。
本研究在平台中共标注了600份病史语料, 同一份语料由两个标注人员同时标注, 对于凡是发现标注不一致的结果, 均由第三个人进行核实并分别与标注人员协商, 确定最终的标注结果。本研究采取了定制化语料标注的方法, 且标注目标具体、简明, 因此在所有标注结果中, 出现不一致的情况较少, 不一致比较集中出现在糖尿病确诊病史的病历中, 例如有些标注人员将病历中出现的“ 考虑糖尿病” 作为糖尿病确诊诊断, 有些则认为不能作为确诊诊断, 最后经过顾问专家讨论, 将其作为次要证据抽取病史, 由具体的抽取数据使用人员确定是否采用。
在解决了标注可能存在的问题之后, 本研究通过自动规则发现以及人工核对结合的方式, 确定了共8条糖尿病史抽取规则, 并针对我们采用的信息抽取处理语言Perl, 对这些规则进行重写后形成实现信息抽取的正则表达式。例如, 病史描述片段“ 既往史:‘ 糖尿病’ 史13年, 未给予治疗未监测血糖, 5年前患脑梗塞, 3年前再发, 未遗留后遗症” , 应用模版“ DM* DT* TIMEUNIT” 的对应正则表达式“ [糖尿病史(.* ?)(年|月|日|天)]” 。部分抽取结果示例见表3。
| 表3 病史抽取结果示例 Table 3 Data extraction results of medical history |
得到可以实际应用的抽取规则库之后, 就可以基于抽取规则库进行病史信息的抽取了。抽取的算法如下所示:①读入一条病史数据D; ②读入一条抽取规则R; ③对数据D应用抽取规则R[简写为R(D)]; ④将抽取结果 S=R(D)并入抽取结果集; ⑤判断规则库是否结束, 若是则⑥, 否则转②; ⑥判断病史数据是否结束, 若是则⑦, 否则转①; ⑦对结果集去重; ⑧结束。
①是指读入一个患者入院记录文档中的现病史、既往史等完整的XML数据片段, 然后通过②~⑤步骤对该段数据逐一使用规则库中的每条规则, 所抽取的结果均进入初始结果表中, 步骤⑥读取下一段XML数据, 并重复上述②~⑤步骤, 整个算法在最后一条XML片段处理完毕后对结果进行去重(步骤⑦)后结束(步骤⑧)。
如果直接利用“ 糖尿病” 这一广义规则, 则所有既往史中类似包含“ 无糖尿病史” “ 无高血压、糖尿病史” 等类似数据都会被错误抽取到糖尿病史中去, 因此, 我们专门对上述明确记录为无病史的情形进行专门抽取并从前面的抽取结果中剔除。与之前的方法类似, 针对糖尿病史的明确否定叙述, 在50个否定叙述的语料条目基础上, 提取出模版“ 否定词* 糖尿病史” 并重写其对应规则“ * (无|否认)(.* ?)糖尿病史” 。
典型的否定描述实例如:“ 既往史:无高血压、心脏病史, 无糖尿病、脑血管疾病病史, 无肝炎、结核、疟疾病史, 预防接种史不详, 无外伤、输血史, 有献血史, 对阿司匹林过敏” 。
病史抽取结果的全面验证是一个较为复杂、耗时的工作, 为了说明抽取结果的有效性, 本研究针对整个抽取结果采用两种验证方法并行的策略。方法一是选取一个科室2015年的全部数据, 进行基于辅助工具的人工验证, 其验证结果为整体抽取结果的指标分析提供参考依据; 方法二是对科室2013年1月至2017年7月所有的数据进行10%取样(其中包含了标注语料所抽样的5%, 原因在于标注语料库实际上是可以直接作为抽取结果验证的金标准, 若抽取结果对于标注语料库的结果都不满足规定指标的话, 则抽取规则库对于其他非标注数据的抽取结果不佳的概率会很大)后进行验证。
进行抽取验证的电子病历数据全部来自住院数据, 本研究依据病案首页数据, 对凡是主诊断、其他诊断中出现糖尿病诊断的患者, 均提取其电子病历中的入院诊断书, 并专门针对病史章节进行糖尿病史信息的抽取及验证。表4显示了科室糖尿病患者病历数据集概况, 其中用于方法一的2015年有效病历共1 436份; 用于方法二的整个科室2013年1月至2017年7月糖尿病患者的10%不放回抽样有效病历1 223份(其中包括所进行抽取语料标注过的600份病历)。
| 表4 验证数据集概况 Table 4 Verification data set |
对于每份抽中被验证的病历, 我们借助平台对病史抽取结果以及包含抽取结果(例如“ 患糖尿病22年” , 或者无糖尿病史结果)的病史文本整体进行对照显示, 并对该抽取结果在原始文本中的位置进行高亮显示, 同时对所有含“ 糖尿” 字样的文本段进行区别显示, 若确认则直接点击确定即可通过并直接进入下一条验证, 研究者4人分两组, 其中一组完成上述1 436份病历中病史抽取结果的核对(每人都独立核对一遍并在最后对两人的核对结果进行一致性检验), 另一组完成上述1 223份病历中(其中600份是标注过的语料, 直接通过程序核对即可确定其抽取结果的正确性, 实际只需核对623份)病史抽取结果的核对, 最终的验证结果见表5。
为使验证结果简明清晰, 表中只对有明确糖尿病病史描述(指同时有糖尿病诊断、糖尿病患病时间)的病历、明确叙述无糖尿病史的病历进行了验证分析。
| 表5 2015年糖尿病病史和全部糖尿病患者10%样本病历抽取结果验证指标 Table 5 Extraction results verification of 2015 EMR data and 10% of all diabetes date |
自然语言处理就是以电子计算机为工具, 对人类特有的书面形式和口头形式的自然语言的信息进行各种类别处理和加工的技术[3]。作为自然语言处理领域的重要分支, 信息抽取是从文本信息中抽取相关数据项的重要方法, 它已经在互联网信息、图书情报分析、通用信息(如地名、人名、时间)等方面取得了显著的研究成果, 应用领域非常广泛[4]。近年来, 随着大数据技术以及人工智能的快速发展, 自然语言处理及其应用形成了新一轮的研究热点。就信息抽取的基本方法而言, 总体可分为两类[5], 第一类是基于规则的抽取方法, 其重点是根据具体数据归纳准确的模版, 根据模版从数据中抽取对应的信息; 第二类是通过命名实体识别(例如识别时间描述、疾病描述)、实体关系推断、关联实体提取并组织到结果表等三个步骤实现信息抽取。这两类方法各有优缺点, 基于规则的第一类方法的优点是简单易用, 缺点是人力耗费严重且对数据实况敏感, 规则通用性差; 第二类方法依赖于丰富、全面的语料库, 通用性较好。
实际上, 在互联网应用快速发展的10年前甚至更早, 半结构化、非结构化数据占比早就超过了传统的结构化数据, 如何利用自动化工具在大量信息中快速、准确地挖掘真正需要的信息, 已经成为研究人员所面临的重大问题。在这种背景下, 产生了信息抽取(information extraction, IE)的研究[6]。但是从整体来看, 信息抽取, 特别是中文语料的信息抽取技术仍有较大的提升空间, 主要面临的问题有信息抽取的局限性、多语言处理成果不显著以及信息抽取的普及性差等。早期的命名实体识别研究工作主要是基于规则, 即对文本中的特定类别的名词进行识别, 研究最多的三种名词是:人名、地名和机构。除此之外, 基于GENIA语料库进行生物医学实体识别研究也迅速发展, 目前主要集中在DNA、RNA、蛋白质等细胞类命名实体的识别方面。
在时间及其相关信息的抽取方面, 已有研究主要是采用基于规则以及基于机器学习方法的信息抽取。基于规则的方法方面, 有研究使用基于多阶段规则的系统确定了“ 入院时间” 或“ 出院时间” 的时间表达式[7], 有研究使用公开的时间提取系统(Heidel-Time)作为基本模块, 并使用专门为临床记录设计的规则来补充Heidel-Time的输出[8]。在基于机器学习的方法方面, 主要是利用机器学习方法, 在精心准备的训练集的基础上首先进行模型训练, 然后应用训练好的模型从文本中抽取需要的信息。常用的机器学习方法有:支持向量机(support vector machine, SVM)、基于最大熵的方法、时间图等。有研究利用时间图将句子进行概括并试图发现有效的模式, 并以此为模版进行信息抽取[9]。机器学习方法的优点在于不需要人类的直觉或者专家经验, 可以重新训练而不需要对任何领域进行重新编程。
随着大数据技术以及机器学习的快速发展, 近年来的相关研究开始使用机器学习方法来提高信息抽取的能力和效率, 例如基于隐马尔可夫模型(hidden Markov model)的命名实体识别方法[10], 根据各类命名实体的构成和用词特点各自指定一套角色标注集, 采用Viterbi算法对切分结果进行角色标注, 再利用隐马尔可夫模型计算其所识别的命名实体的输出概率。
在医学自然语言处理领域, 尤其是临床电子病历数据的研究应用方面, 近年来受到比较广泛的关注, 出现了较多有应用价值的成果[2], 例如基于电子病历信息抽取的诊断推荐[11], 帮助全科医生、护士等专业人员改进临床数据的收集与整理[12, 13]。同时, 由于很多电子病历包含非结构化的自由文本, 病历中很多文本中的内容并没有记录到结构化的编码数据表中, 因此, 仅使用结构化数据进行病历检出时, 检出的效率比较低, 将结构化的数据与非结构化的自由文本结合起来可以提高检出效率。有研究报道采用自然语言处理的方法从电子病历中提取所有糖尿病患者的相关人口和诊疗信息, 用以确定患者的准确数量及临床诊疗关键信息[14]; 通过抽取2型糖尿病患者的临床风险指标数据用以研究模型并尝试预测寿命[15]; 应用自然语言处理技术来进行癌症患者的识别[16]; 从电子病历中抽取患者高血脂症、高血压和肥胖症等症状或诊断信息, 临床的生化指标以及家族史、吸烟等危险因素, 以此为基础信息建立辅助医生进行临床诊疗的疾病风险预测模型[17]。
综合目前信息抽取的研究及应用工作, 总体而言, 基于规则的抽取以及基于实体及其关系识别这两类方法互为补充[7], 且在一些基础性研究方面逐渐趋于融合[18]。例如本研究采用的规则抽取方法中的规则归纳, 其基础就是需要建立平衡、全面的专用标注语料库, 而与此同时, 该语料库可以直接作为第二类方法进行时间、疾病实体识别的基础语料库。反过来, 通过命名实体识别技术, 可以在文本中发现一些名词的同义词, 进一步丰富信息抽取的模版与规则。
就本研究中的糖尿病史抽取, 为了避免叙述过于繁琐, 只对病历文本数据中明确有糖尿病确诊以及对应时间描述的病史进行了识别和抽取。实际上, 我们将糖尿病确诊的证据分为三个级别:最高级别是有明确的糖尿病确诊以及病史时间叙述, 为1级; 接下来是有明确的生化指标, 主要是空腹血糖水平、饭后2 h血糖水平、随机血糖水平以及对应的指标获取时间的叙述, 为2级; 最低水平的证据是服用二甲双胍等药物及其对应的服用日期时间点的叙述, 为3级。最终抽取的结果表中, 若有多个级别的证据同时存在且时间信息一致, 则只保留最高级别证据的抽取结果, 同时, 整个病史抽取结果中, 对于同一患者的不同时间点的证据均予以保留, 由具体的临床研究分析人员自行决定取舍。
需要说明的是, 糖尿病史的抽取对于总体数据而言, 除了上述有明确糖尿病史的病例, 另一部分就是首次确诊的糖尿病(否定检出所确定的糖尿病患者即属于首次确诊的情形)的病史数据提取较为简单, 我们通过直接提取电子病历中的入院日期即可。同时, 在实际抽取中, 对于部分病史描述出现“ 有糖尿病史” 却没有明确确诊时间点的患者, 我们也会生成病史结果的参考列表, 一方面可以通过上述2、3级的证据进行补充, 另一方面方便提示临床医生在患者后续就诊时尽量补充完整。最后, 对于多次入院治疗的糖尿病患者而言, 提取的病史会出现重复的情形, 例如某患者在第一次入院时的病史提取结果为[糖尿病, 20年前], 而2年后入院的电子病历数据提取结果为[糖尿病, 22年前], 这种情况则应以最近一次入院的电子病历数据中所提取的结果为准。
本文主要采用自然语言处理与基于规则的信息抽取方法结合, 实现从非结构化的电子病历文本数据中抽取定制化的信息, 并与Stanford的中文自然语言处理包[19](以下简称CoreNLP)的方法进行实验比对, 本研究方法在糖尿病的识别上要优于CoreNLP, 其原因一方面很大可能在于CoreNLP方法对训练语料库的依赖很强, 而在实际应用中, 其所依赖的标注语料库很难完全覆盖所有的可能, 因此相对于本文采取的基于多层规则(简单规则实际上已经简化为单个疾病的搜索)的抽取方法, 其召回率会有所不足; 另一方面, 数据中标准术语的总体符合程度对于方法的各项指标会有较大影响, 越是符合标准术语的数据, 其在通用方法及软件包的处理效果越好。总体而言, 作为有代表性的自然语言处理工具, 在实际应用将其作为抽取结果的基线准则, 会对抽取方法的改进、各项指标的提升有很大的益处。
同时可以看到, 语料库无论在基于规则的数据抽取还是句型发现等机器学习方法中, 都起着举足轻重的作用, 但目前电子病历语料库及其标注过的可用的金标准语料非常少见, 我们认为这是目前电子病历数据中非结构化数据的有效利用的不利因素。在医院作为应用主体, 目前没有足够的人力和资源进行大量全面的语料库建设的情况下, 我们建议通过定制化语料库建设及众包(crowd outsour-cing)标注的方法来逐步解决这一问题, 边建设、边应用, 多次迭代, 逐渐建成符合中文电子病历自然语言处理的基础语料库及知识库。
后续工作我们考虑在结合已有方法, 应用机器学习算法实现基于标准术语的语料自动标注、实体识别以及实体关系的基础上, 实现较高效率的定制化信息抽取与临床分析应用。
(志谢:郑州大学第一附属医院内分泌科, 北京大学基础医学院的谢双莲、宋凯等在本研究工作中提供很多帮助, 在此深表感谢!)
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|