1 资料与方法
1.1 数据来源
1.1.1 基因甲基化、基因表达和循环蛋白质的数据来源
1.1.2 PCa队列的数据来源
1.2 SMR方法
1.3 共定位分析
1.4 多组学证据分级依据
1.5 细胞实验
1.5.1 细胞培养和转染
1.5.2 细胞计数
1.5.3 细胞划痕实验
1.5.4 Transwell侵袭实验
1.5.5 蛋白免疫印迹实验
2 结果
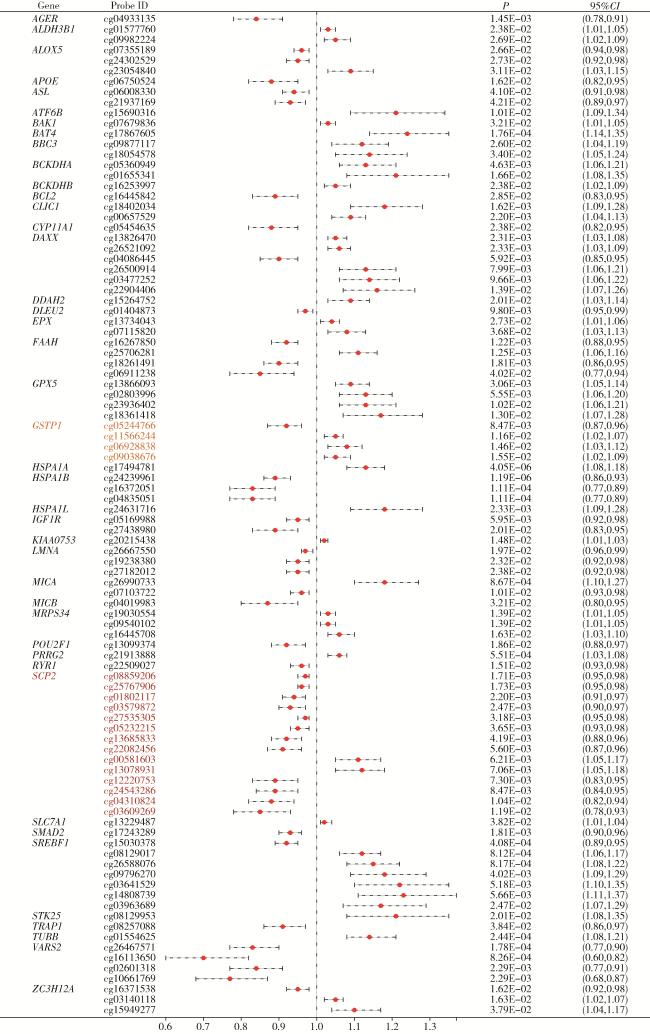
2.1 氧化应激相关基因甲基化与PCa
图1 基于汇总数据的孟德尔随机化分析中氧化应激相关基因甲基化水平与前列腺癌的关系Figure 1 Association between methylation levels of oxidative stress-related genes and prostate cancer risk in summary-data-based Mendelian randomization (SMR) analysis |
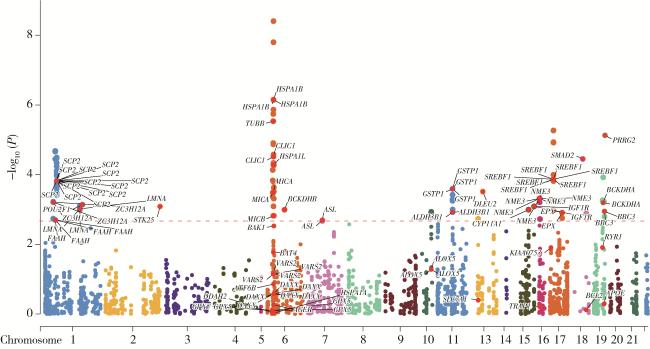
2.2 氧化应激相关基因表达与PCa
图3 不同氧化应激相关基因表达与前列腺癌的关系Figure 3 Association between the expression of oxidative stress-related genes and prostate cancer A, relationship between the expression of oxidative stress-related genes and prostate cancer in Mendelian randomization analysis; B, Manhattan plot of oxidative stress-related gene expression with statistically significant associations. |
表1 GTEx数据库中与前列腺癌具有显著关联的氧化应激相关基因Table 1 Oxidative stress-related genes significantly associated with prostate cancer in the GTEx database |
| Gene | OR | 95%CI | P |
| COX6B1 | 1.16 | 1.059-1.261 | 0.032 |
| BAK1 | 0.95 | 0.915-0.981 | 0.047 |
| TXN | 1.13 | 1.041-1.218 | 0.047 |
| PLA2G2A | 1.05 | 1.015-1.081 | 0.047 |
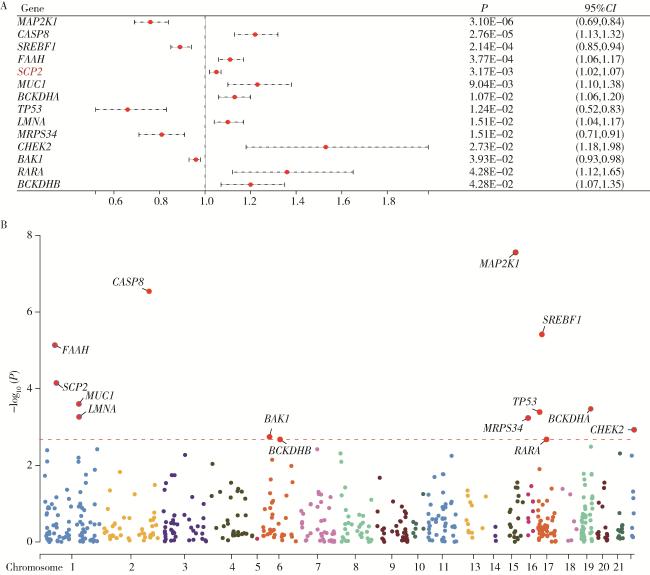
2.3 氧化应激相关循环蛋白质与PCa
图4 不同氧化应激相关基因循环蛋白质层面与前列腺癌的关系Figure 4 Association between circulating protein levels of oxidative stress-related genes and prostate cancer A, relationship between oxidative stress-related circulating proteins and prostate cancer in Mendelian randomization analysis; B, Manhattan plot of oxidative stress-related proteins with statistically significant associations. |
表2 deCODE数据库中与前列腺癌具有显著关联的循环蛋白质Table 2 Circulating proteins significantly associated with prostate cancer in the deCODE database |
| Protein | OR | 95%CI | P |
| LPO | 0.97 | 0.947-0.983 | 0.009 |
| HSPA1A | 0.97 | 0.948-0.983 | 0.009 |
| PLA2G2A | 1.03 | 1.011-1.053 | 0.049 |
2.4 氧化应激相关基因与PCa的多组学研究结果
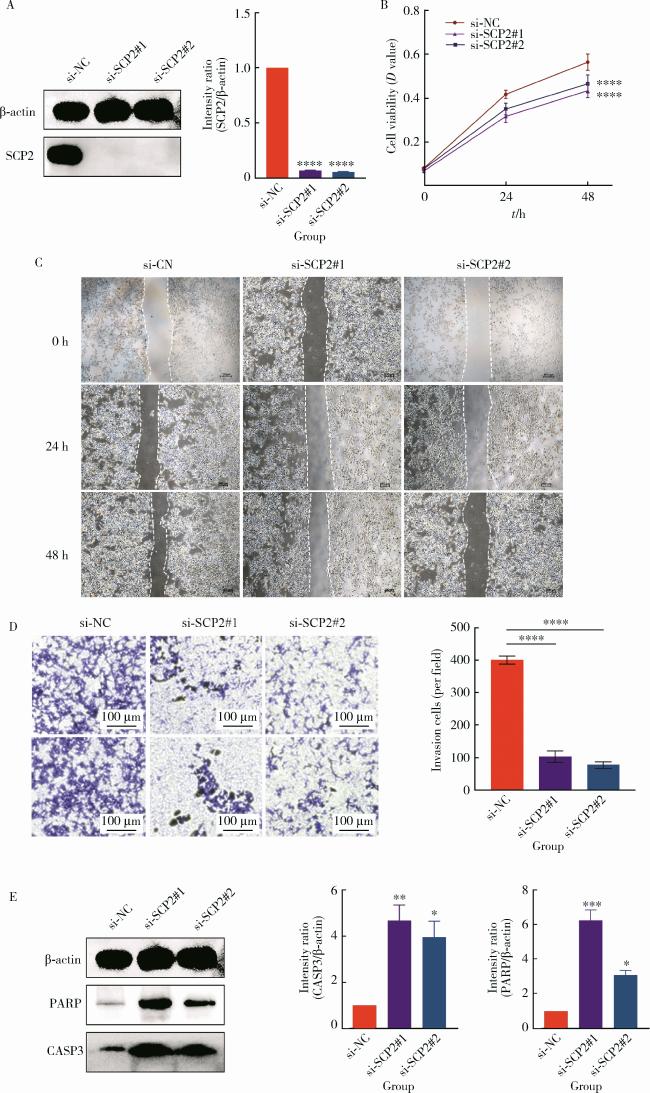
2.5 敲除SCP2可抑制PCa肿瘤细胞的致癌表型并促进其凋亡
图5 敲除SCP2可抑制前列腺肿瘤细胞的致癌表型并促进其凋亡Figure 5 Knockdown of SCP2 suppresses the oncogenic phenotype of prostate cancer cells and promotes apoptosis A, siRNA treatment of C4-2 cells significantly decreased SCP2 protein expression (* * * *P < 0.000 1); B, knockdown of SCP2 significantly reduced the proliferative capacity of C4-2 cells (* * * *P < 0.000 1); C, D, knockdown of SCP2 markedly suppressed the migration and invasion abilities of C4-2 cells (* * * *P < 0.000 1); E, knockdown of SCP2 increased the expression of apoptotic markers CASP3 (* *P < 0.01 & *P < 0.05) and PARP (* * *P < 0.001 & *P < 0.05) in C4-2 cells. |